Services
서비스
NGS
Amplicon Metagenomic Sequencing
Amplicon metagenomic sequencing은 주어진 샘플 내 존재하는 박테리아, 균류 등을 식별 및 비교하는 효율적이고 경제적인 시퀀싱 방법입니다.
NGS 기반 분석은 배양이 어렵거나 불가능한 복잡한 마이크로바이옴 또는 환경 내 군집다양성의
계통 분류학적 비교를 가능하게 하며 시각적, 통계적 지표를 제공함으로써 연구자의 이해를 돕습니다.
Universial primer set을 사용한 Amplicon sequencing이 아닌 경우에는 충분한 컨설팅을 거쳐 실험 및 분석이 진행됩니다.
적용 가능 연구
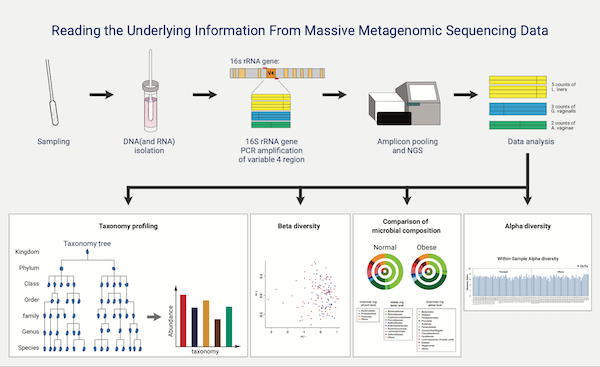
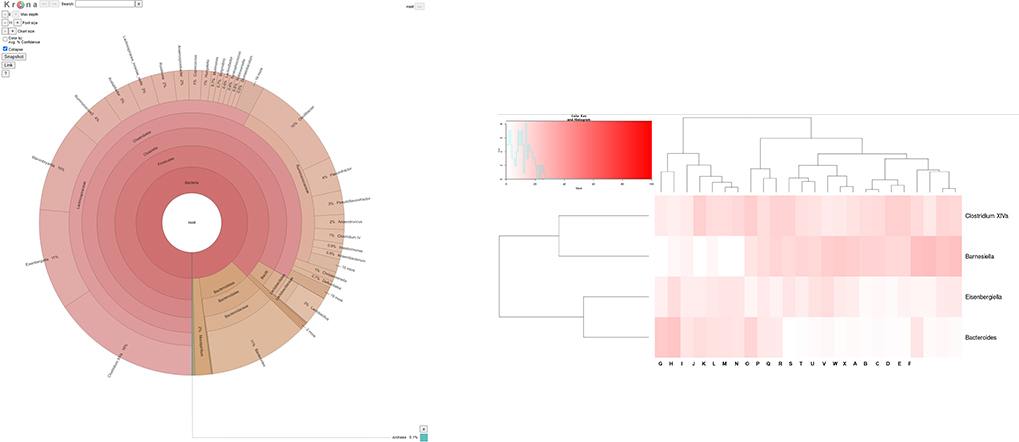
- Taxonomic profiling
- Comparision analysis of microbial community
- Diversity statistics (alpha-, beta- diversity)
샘플&실험정보
| Sample Requirement | gDNA > 100ng (minimum 10ng) 20ng/µl DIN > 6 |
|---|---|
| Library Kit | Illumina |
| Sequencing Platform | Illumina MiSeq |
| Recommended Sequencing Depth | Up to 50,000 reads per sample |
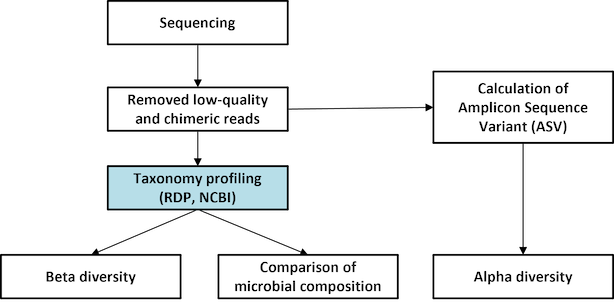
생명정보학 분석
- Workflow
-
Standard Analysis
Pick De novo OTUs
Microbioal taxonomic Profiling
Diversity statistics(alpha-, beta- diversity)
Reference
[1] Lim, M. Y. et al., (2020). Changes in microbiome and metabolomic profiles of fecal samples stored with stabilizing solution at room temperature: a pilot study. Scientific reports, 10(1), 1789.
[2] Gunathilake, M. N. et al., (2019). Association between the relative abundance of gastric microbiota and the risk of gastric cancer: a case-control study. Scientific reports, 9(1), 13589.
[3] Hu, H. J. et al., (2015). Obesity Alters the Microbial Community Profile in Korean Adolescents. PloS one, 10(7), e0134333.
[4] Saha, S. et al., (2019). Microbial acclimatization to lipidic-waste facilitates the efficacy of acidogenic fermentation. Chemical Engineering Journal, 358, 188–196. https://doi.org/10.1016/j.cej.2018.09.220
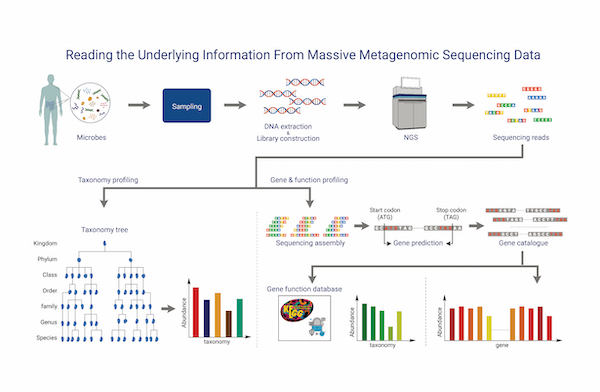
Shotgun Metagenomic Sequencing
NGS 기반 Shotgun metagenomic sequencing은 배양이 불가능한 미생물 혹은 복잡한 환경에 존재하는 모든 유기체의 유전자를 포괄적으로 검출할 수 있으며,
미생물 다양성, 기능적 특징을 제공함으로써 환경 내 미생물 간 관계, 환경과 미생물의 관계 등 이해를 돕습니다.
시료 내 존재하는 모든 유기체를 Contig 레벨로 조립하여 제공하며, Genome 상의 CDS region 등을 예측한 후
UniProt, RefSeq, Pfam 등의 데이터베이스를 활용하여 유전자 Annotation을 진행합니다.
적용 가능 연구
- Identification of microbial community diversity in the environmnet
- Metagenome assembled genomes
샘플&실험정보
| Sample Requirement | gDNA > 10µg (minimum 5µg), 100ng/µl, DIN > 8 |
|---|---|
| Library Kit | Illumina |
| Sequencing Platform | Illumina NovaSeq6000, PacBio Sequel Ⅱ |
| Recommended Sequencing Reads | 70 million read pairs per sample |
생명정보학 분석
- Workflow
-
-
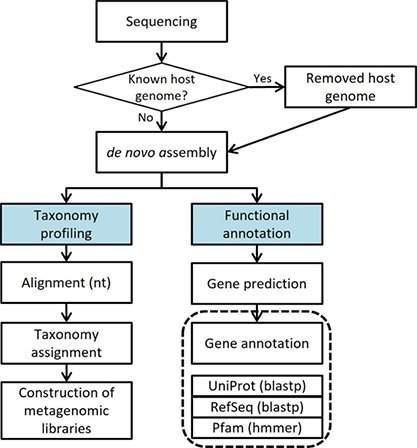
Standard Analysis
Remove host sequences
Metagenome assembled genomes
Prediction genomic region
Gene annotation
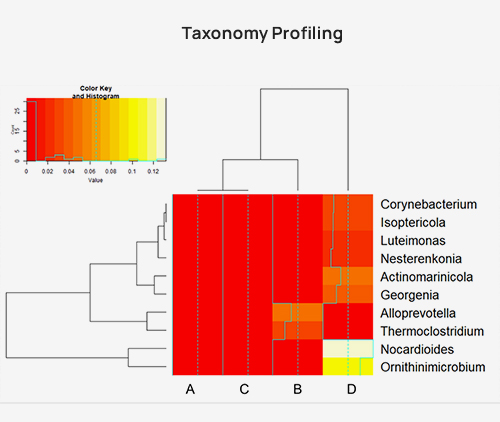
Taxonomy assign
-
Advanced Analysis
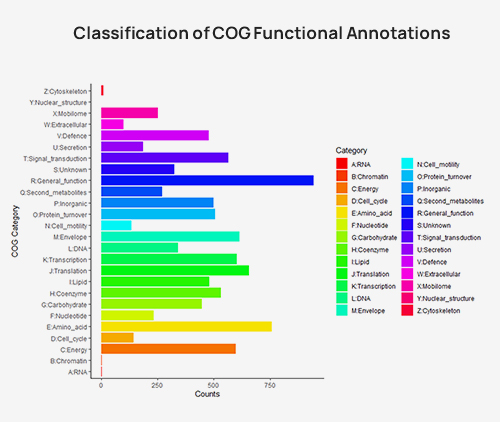
Classification of COG functional annotations
No content
No content
No content
No content
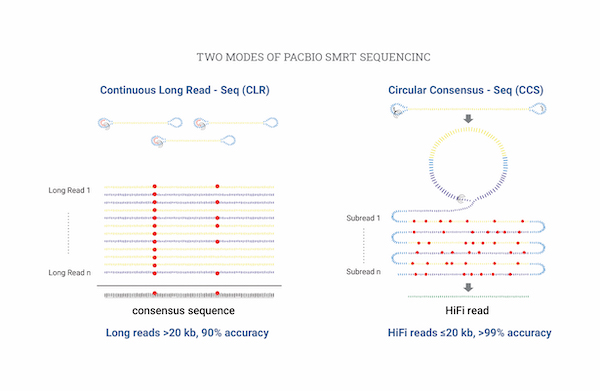
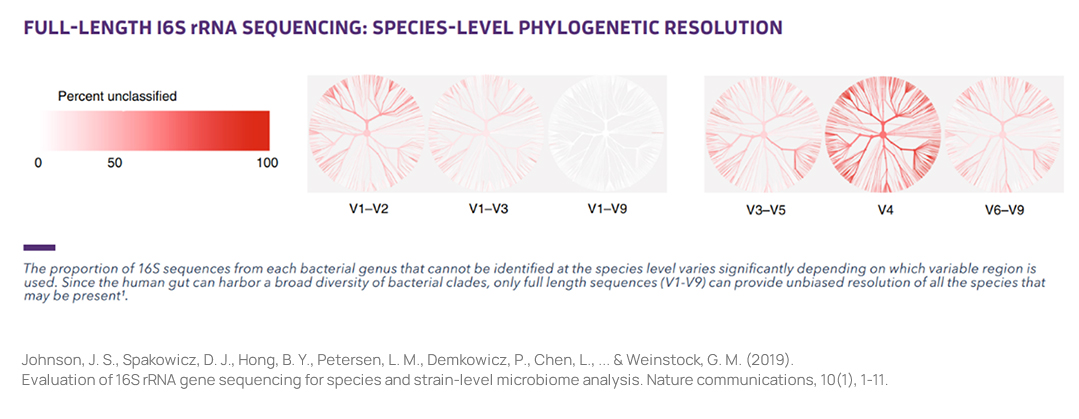
Full Length 16S rRNASequencing
NGS 기반 마이크로바이옴 연구는 비용 부담이 적은 대신 판독 영역이 짧아 종 수준에서 오분류하는 것이 큰 과제로 남아있습니다.
PacBio의 Single molecule, real-time(SMRT) sequencing은 16S rRNA 및 18S rRNA 영역에서 약 1.5Kbp를 해독하여
종 수준에서 높은 정확도로 수준 높은 해상도의 프로파일링 결과를 제공합니다.
적용 가능 연구
- Microbial taxonomic profiling
- Full length 16S rRNA genomic analysis
샘플&실험정보
| Sample Requirement | gDNA > 1µg (minimum 5µg), 100ng/µl, DIN > 8 |
|---|---|
| Library Kit | PacBio |
| Sequencing Platform | PacBio Sequel Ⅱ |
| Recommended Sequencing Depth | - |
생명정보학 분석
- Workflow